搜索

蛋白質是結構復雜的生物大分子,其表面無規分布著多個疏水相互作用⛹🏿、靜電相互作用位點👸🏼,很難對其自組裝過程進行有效控製以獲得結構精確、形貌規整的組裝體。這限製了蛋白質組裝材料在生物醫學、傳感器件等方面的應用🏃🏻♀️➡️。目前👩🦯➡️,常用的調控蛋白質表面相互作用的方法是通過重組表達獲得氨基酸序列突變的蛋白質🍿,進而實現蛋白質自組裝等👏🏽。但是🌂,重組表達的方法存在過程相對繁瑣、蛋白質擴量困難的缺點🤾🏽♂️,且這類蛋白質自身的結構和對稱性是所得組裝體結構和形貌的重要決定因素⛅️,很難利用一種蛋白質獲得具有不同形貌和結構的組裝體。
為了解決上述問題,我系陳國頌-江明課題組提出👩🏿🚒:利用蛋白質自身具有的寡糖結合位點,向天然蛋白質中引入經巧妙設計的小分子誘導配體,以此建立和控製蛋白質之間的超分子相互作用,實現了天然蛋白質的精確自組裝。據此,先後獲得了三維框架結構晶體和具有螺旋結構的微米管🤦🏿,相關成果前期發表在Nat. Commun.和J. Am. Chem. Soc.上。
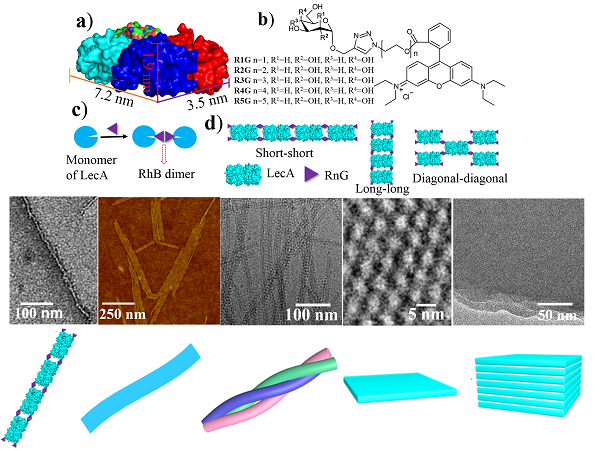
最近,他們發現通過簡單地調節誘導小分子結構中糖與羅丹明之間連接基團的長短RnG (n= 1, 2, 3, 4, 5)🔂,即可對具有長方體結構的蛋白質LecA的精確自組裝進行有效地調控👩🏿🍼,從而簡單、方便地製備一維納米線、納米帶、二維晶體片層、以及三維片層等蛋白質組裝結構👨🏻🏫。他們通過冷凍透射電鏡技術,對於蛋白質在上述組裝體內的排列進行了清晰的表征👏🏻。為了從理論上對於蛋白質自組裝的結果進行解釋和預測,他們與蘇州大學和南京大學的研究小組合作🤦🏼♂️,利用全原子模擬和布朗動力學模擬分別從微觀和介觀層次分析了小分子結構對蛋白質自組裝的調控作用,發現理論模擬與實驗結果可以很好地吻合。相關研究成果已在線發表在Angew. Chem. Int. Chem.上 (Guang Yang‡, Hong-ming Ding‡, Zdravko Kochovski‡, Rongting Hu, Yan Lu, Yu-qiang Ma*, Guosong Chen*, Ming Jiang, Highly Ordered Self-Assembly of Native Proteins into 1D, 2D, and 3D Structures Modulated by the Tether Length of Assembly-Inducing Ligands. DOI: 10.1002/anie.201703052), 並被選為Hot Paper。沐鸣开户、聚合物分子工程國家重點實驗室為第一單位🀄️,我系博士生楊光⚓️,蘇州大學丁泓銘博士以及德國海姆霍茲柏林軟物質研究所的Zdravko Kochovski博士為共同第一作者😈,馬余強與陳國頌為共同通訊作者🧑🍳🫱🏼。上述研究得到了國家自然科學基金委的資助。